Jak objaśniają w swojej publikacji, która trafiła na łamy Nature Biotechnology, to nie pierwsze podejście do tematu. Nowa baza danych, czyli AGORA2, wykorzystuje bowiem informacje zebrane w czasie pierwszej próby, zakończonej powstaniem bazy AGORA1.
Czytaj też: Ten smartwatch jest napędzany żywym organizmem. Trzeba go karmić, bo inaczej przestanie działać
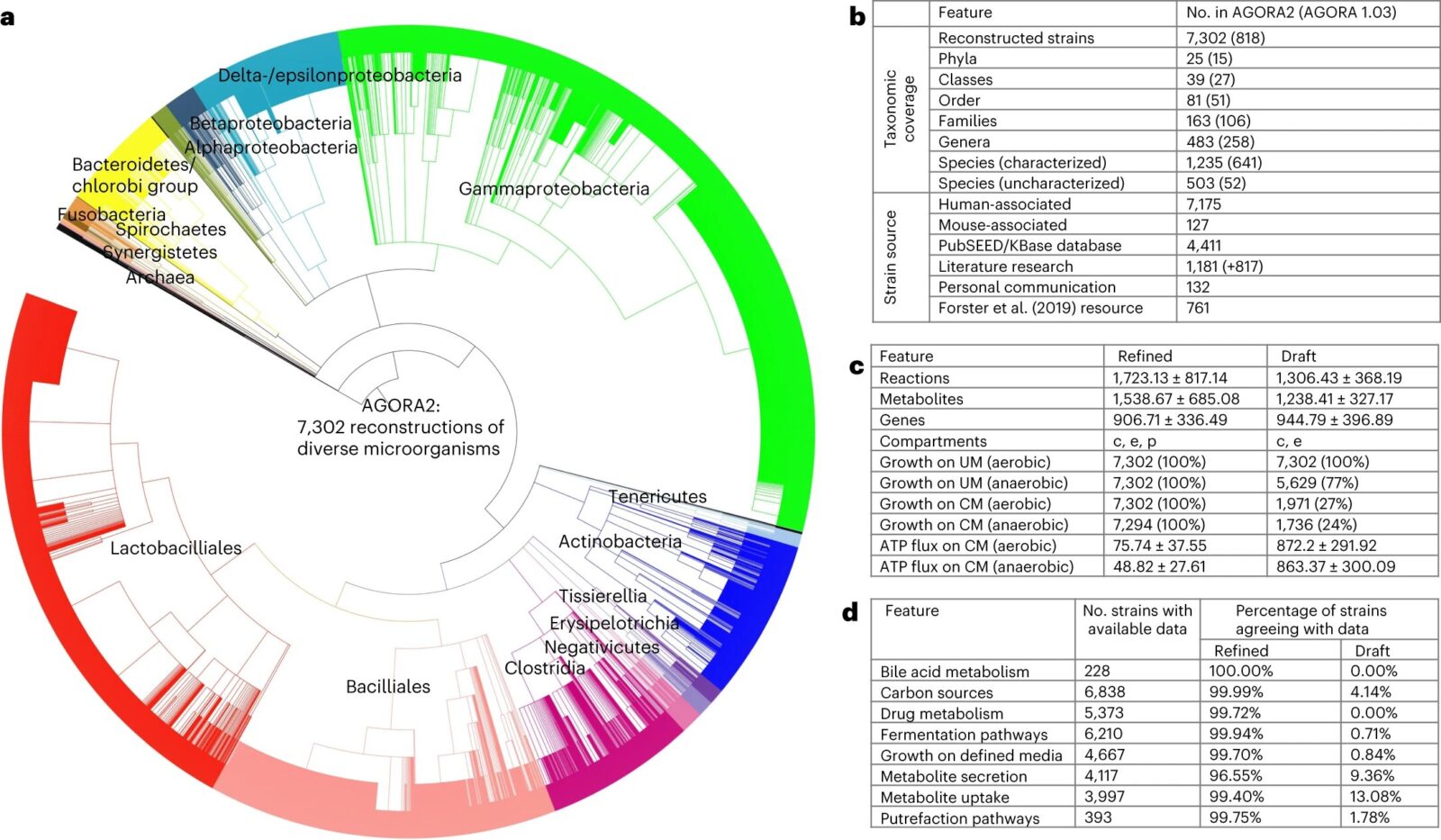
Ta najnowsza wersja składa się z dokładnie 7203 cyfrowych mikrobów, które powinny być szczególnie przydatne w kontekście badań na metabolizmem leków. Innymi słowy, autorzy stworzyli podwaliny dla systemu pozwalającego na testowanie różnego rodzaju terapii oraz badanie reakcji pacjentów na takowe.
Wśród konkretnych przypadłości naukowcy wymieniają przede wszystkim nieswoiste zapalenie jelit, chorobę Parkinsona i raka jelita grubego. Jak wyjaśnia Ines Thiele, która stała na czele zespołu zajmującego się badaniami w tej sprawie, AGORA2 jest kamieniem milowym w kierunku symulacji komputerowych umożliwiających analizę interakcji na linii człowiek-mikrobiom-lek w kontekście zastosowań z zakresu medycyny precyzyjnej.
Ludzie są gospodarzami niezliczonej ilości mikrobów. Podobnie jak my, mikroby te odżywiają się i wchodzą w interakcje ze swoim środowiskiem. Biorąc pod uwagę, że wszyscy jesteśmy unikalni, każdy z nas gości indywidualny mikrobiom i oczekuje się, że nasz metabolizm również będzie się różnił między jednostkami. […] Oprócz naszego pożywienia, nasze indywidualne mikrobiomy metabolizują również leki, które przyjmujemy. Ten sam lek może zatem przejawiać zróżnicowane efekty u różnych osób z powodu różnic w metabolizmie wykonywanym przez poszczególne mikrobiomy. wyjaśnia autorka
Symulacja komputerowa AGORA2 składa się z 7203 cyfrowych mikrobów
Do jakich wniosków zdążyli już dojść autorzy? Przede wszystkim odnotowali, iż metabolizm leków różni się u poszczególnych osób i to często na zaskakująco dużą skalę. Przyczyną takiego zjawiska mogą być właśnie różnice w mikrobiomach. Praca badawcza wskazuje także na możliwość identyfikacji mikrobów i procesów metabolicznych dla wybranych leków skorelowanych z obserwacjami w warunkach klinicznych.
Czytaj też: Naukowcy opanowali zmysł dotyku. Kolejny krok do stworzenia symulacji idealnej
W eksperymentach wzięło udział 616 osób chorujących na raka jelita grubego oraz przedstawicieli grupy kontrolnej. Jak się okazało, baza danych AGORA2 dostarcza spersonalizowanych informacji opartych na przewidywaniu potencjału konwersji leków w mikrobiomie jelitowym. Mikrobiom pacjentów i zdrowych osób był skorelowany z wiekiem, płcią, wskaźnikiem masy ciała i stadiami choroby. Co to oznacza w praktyce? Że naukowcy, mając do dyspozycji nowe narzędzie, mogą tworzyć cyfrowe reprezentacje i prognozy przeznaczone dla rozbieżnych mikrobiomów.